【实验技术笔记】利用重组载体做基因过表达(pCDH载体)
时间:2022-10-27 17:30:00
文章目录
- 1. 构建基因过表达载体
-
- 1.1 设计 PCR 引物
- 1.2 PCR 扩大目的基因
- 1.3 酶切载体和 PCR 产物
- 1.4 电泳并回收酶切割产品
- 1.5 连接
- 1.6 转化
- 1.7 选择阳性克隆并进行鉴定
- 2. 转染
- 3. 测试表达效果
- 附表:常用酶切位点保护碱基
为什么要做基因表达操作?
- 探索影响细胞表面变化(增殖、凋亡、焦亡等)的分子,了解这些分子之间的关系(机制)
- 分子-机制-表型之间的关系
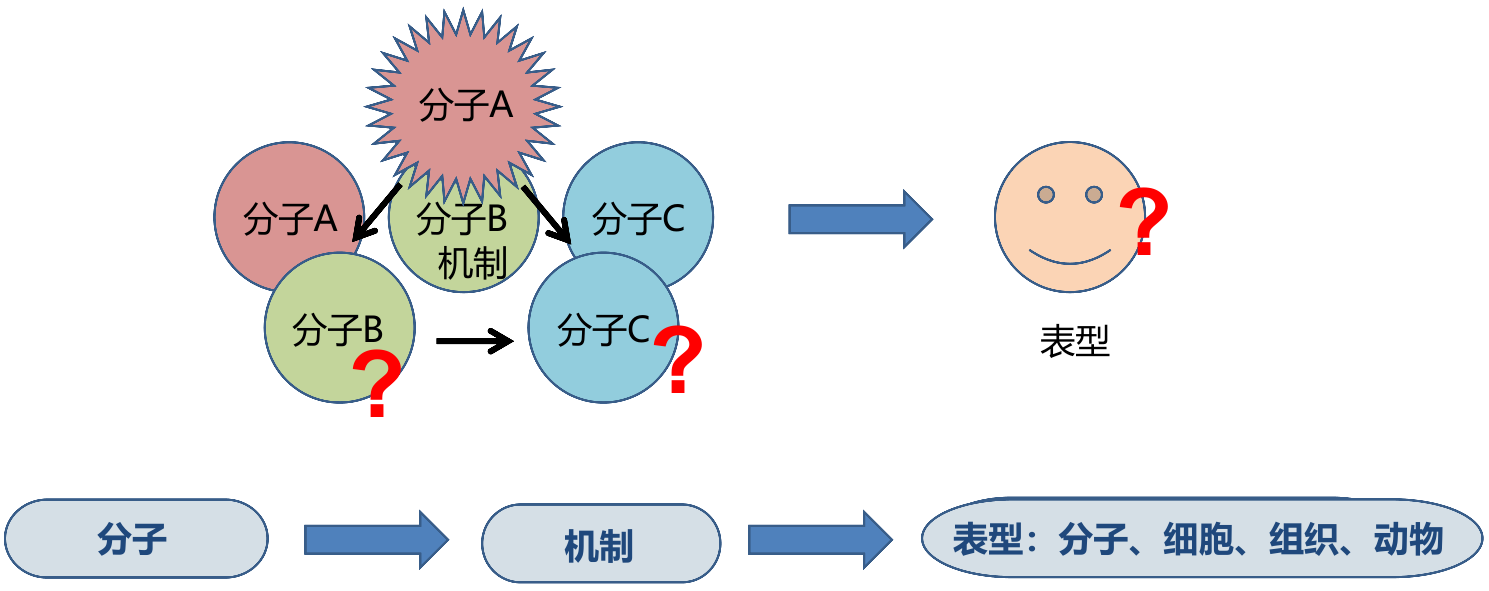
- 对谁操作?
① DNA,如 CRISPR-Cas9 细胞本身 DNA 删除、添加或替换;最常见的操作如过表达,将外源 DNA 引入细胞,用细胞的转录翻译系统表达相应的蛋白质。
② 利用具有干扰作用的小干扰 RNA,降解细胞内源 RNA 或抑制翻译,从而影响蛋白质的表达。
操作过程中基因过表达
1. 构建基因过表达载体
1.1 设计 PCR 引物
- 参考:载体构造实例分析-构造 SETD3-pEGFP-N1(Snapgene 设计引物)
(1)寻找 CDS 序列
- 以 TNF 为例,将 CDS 序列复制到 DNAMAN,保存为 .seq 文件。
(2)分析酶切位点:找出载体多克隆位点 MCS 是的,酶切位点插入基因中没有。
- DNAMAN 中载入 TNF 序列文件:Sequence → Load Sequence → From Sequence File → 选择保存在前面的 TNF.seq 序列文件。
- 寻找 TNF 基因中的酶切位点:Restriction → Restriction Analysis → 勾选 Show summary 和 Show sites on sequence → 下一步→Select All → 完成。
- 注意输出结果
Non Cut Enzymes(不会切 TNF 基因酶切位点),找出载体(以pCDH-CMV-MCS-EF1-copGFP以载体为例,多克隆位点 MCS 是的,酶切位点插入基因中没有。
- 找出切割效率高的酶切位点:达到切割效率 90%以上是最好的;排除同尾酶,即切割后性末端不能相同(如 XbaⅠ和 NheⅠ是同尾酶)。常用酶切位点表(含保护碱基)
- 找出在相同 buffer 和温度下工作的酶切位点:
① 利用 NEB 官网的Double Digest Finder工具,输入选取的在载体上不相邻 双酶切位点(如XbalⅠ、BamHⅠ),查看酶的Buffer、反应温度及相应的酶切效率。
如图,BamHⅠ标记了 星号,表示酶在对应 buffer 有星号活性(非特异性酶切活性),酶会乱切,把 DNA 切断,所以要避免有星号活性的酶,此处 BamHⅠ不可选。
② 但在输入 BamHⅠ时提示还有 BamHⅠ-HF, HF 代表是转化后的切割效率较高,选择 BamHⅠ-HF 结果如下图所示,XbalⅠ和BamHⅠ-HF都能在 37℃ rCutSmart Buffer 中达到 酶切效率100%,可选。
(3)添加保护碱基和 Kozak 序列
- 在酶切位点外侧添加保护碱基,可为外切酶提供支撑点,提高酶切效率。
XbalⅠ和BamHⅠ-HF保护碱基分别是 GC 和 CG - Kozak 序列(GCCACC AUG G)是真核生物 5’帽子结构后面的核酸序列可以与翻译的起始因素相结合。介导包含 5’帽结构 mRNA 翻译的开始。对应原核生物 SD 序列。核糖体可以识别这个序列,并作为翻译的起点。
- 注意:下游引物是规则框架设计 反向互补序列。
(4)验证引物是否合适 PCR: (不合适也没办法,载体引物的局限性),只看部分与目的基因互补(目的基因 CDS 序列),引物 Tm 值只计算与目的基因完全互补的部分。
- 参考:载体构造实例分析-构造 SETD3-pEGFP-N1(Snapgene 设计引物)
1.2 PCR 扩大目的基因
- 参考:【实验技术笔记】RNA 抽提 反转录 PCR PCR 引物设计 RT-qPCR
- 与做 RT-PCR 只是酶不同。
- 推荐:高保真酶 TOYOBO
KOD-Plus-Neo(KOD-401)便宜的大碗,性能好。
Agilent PfuUltra II Fusion HS DNA Polymeras(600670)稍微贵一点,性能强。 - 反应液配制:配置后,可额外添加
0.5 μl ExTaq,加 ExTaq 的 PCR 产品尾带突出 A 碱基,做起来方便 TA 克隆。
- PCR 程序:克隆 PCR 建议用 Touchdown PCR 程序设置。在普通 PCR 前加 10 个循环的 Touchdown 程序,一般从 68℃降到 58℃(如果还 P 不出来,可以从 72℃开始降到 58 或 55℃)。
1.3 酶切载体和 PCR 产物
- 酶切体系:分 2 管,一管切载体,一管切载体 PCR 产品。各种内切酶 1ul,10×buffer 2ul,37 ℃ 酶切 2h 后跑胶回收。
- 推荐: NEB的酶,选择多,高效的酶多。
1.4 电泳并回收酶切割产品
- 1.1%琼脂糖凝胶电泳后分别切胶回收载体片段和目的基因片段,0.5cm 胶块 300μl 熔胶液,55-60℃ 加热熔胶。
- 2、上柱,离心 5000g×5min 过柱。
- 3、700ul wash buffer,最大速离心 1min 洗柱 2 次,最大速离心 2min。
- 4、50ul ddH2O(最好加热到 5060℃),最大速离心 2min 洗脱(建议洗脱 2 次,把第一次洗脱的液体吸到柱子里再离心一次,提高产率),Nanodrop 测浓度。
- 推荐:胶回收试剂盒,OMEGA,
Gel Extraction Kit(D2500-01)不推荐国产的如天根、生工(效率较差)。
1.5 连接
-
用
T4 DNA Ligase把 PCR 产物酶切回收片段 (insert) 和载体酶切回收片段 (vector) 连接在一起,16℃连接 4h 或过夜。(连接温度以 16℃为佳,但是 16℃的连接温度没有金属浴不好办,还是 4℃
过夜(12-16h)比较方便,其实室温下几个小时也是可以的。) -
连接体系:
◆ 载体酶切回收片段加到 100ng 以上,加少了连接产物可能比较少;
◆ 连接体系按连接酶说明书配制(通常 10μl 连接体系中 T4 DNA 连接酶加 1μl,10×buffer 加 1μl)
◆ 酶切载体片段与目的片段的摩尔比例,建议以 1:5-1:10 之间比较合适,可使用 克隆构建摩尔比计算工具 进行计算每 μl 酶切载体片段需要加多少 μl 目的片段,将 ABCD 4 个位置的示例数据替换成你的实验数据,即可自动计算出不同比例下每 μl 酶切载体片段需要加多少 μl 目的片段。(可以 vector 加 100ng,然后剩下的体系全部加 insert,不加水?)
-
推荐:NEB,
T4 DNALigase(M0202)
1.6 转化
- 1、从-80℃冰箱中取出感受态细胞(
Top10感受态或DH5α感受态等),置于冰盒上解冻,并做好标记; - 2、连接产物 10 μl,加入到 20μl 感受态中;(天根的感受态细胞一支 100ul,可以不按说明书全用掉,可以分装成 20ul 来用,后面加新鲜 LB 时也按比例缩小)
- 3、混匀后冰浴 30min;
- 4、42 ℃ 热激 1min30s;(此步可换成室温静置 5-15min,一般冬天静置 15min,夏天 5min,效率几乎相同);
- 5、冰浴 3-5min;(省略此步,依旧实验顺畅,但是建议加上,感觉对效率还是有一定影响的);
- 6、加 100ul 新鲜无抗生素 LB 培养基,37 ℃ 200rpm 摇床上复苏 1h;
- 7、铺板:涂布含抗生素的 LB 平板,37℃倒置培养 16h。(在生物安全柜中,将摇菌完成的产物加入到 LB 平板中,尽量加在中间,不要加在边上,用涂布棒涂布均匀;37℃正置培养 1h,待液体稍干后倒置培养 12-16h)
- 推荐:TIANGEN,
TOP10 感受态细胞(CB104)天根的都可以 - LB 培养基配方:
- LB 平板制备:
① 配 LB 琼脂:有配方,但是建议直接买预混好的,按说明书配,如百思的 BS1080。融解过程需要适当加热,最好用可加温的磁力搅拌器,一般 65℃即可完全融解。
② 融解完全后,倒入干净的容器(如高压过的丝口瓶),高温高压灭菌。
◆ LB 培养基高压之后会有少许沉淀,属正常现象,摇菌过程中会自行溶解。如果不溶解,则要怀疑是不是培养基污染。建议摇菌前将培养基摇匀后再添加到摇菌管、离心管或三角瓶中。
③ 高压后的 LB 琼脂,冷却到 50℃左右,加入相应的抗生素,如卡那霉素,氨苄青霉素等,混匀。若是已经冷却完全的 LB 琼脂,建议使用微波炉,开小火,每次加热 1min,直到完全融化为止。
④ 取无菌的细菌培养皿,趁热将加抗生素的 LB 琼脂倒入培养皿,一般 90mm 皿倒 10ml(二分格的皿,一边倒 5ml,不好把握时,可用移液器)。
⑤ 倒入 LB 琼脂的板子,放平,冷却,待 LB 琼脂凝固,保鲜膜包裹,存放 4℃。
1.7 挑选阳性克隆并鉴定
-
1、挑阳性克隆摇菌:用无菌枪头挑阳性克隆到含 1‰ 抗生素的 LB 培养基(大肠杆菌)中,37℃ 200rpm 摇床上培养过夜(10-12h,不要超过 16h,时间过长,菌长老了提取效果就不好。当然如果给的培养基量大,可以适当延长摇菌时间。);
-
2、菌落鉴定:
◆ 方法一:酶切鉴定:摇好的菌液抽提质粒(小抽),做酶切、电泳,看目的片段有没有插入载体中(先摇菌,再提质粒,酶切完跑胶鉴定,好麻烦。而且很费钱呀,限制性内切酶、质粒小提的盒子,都不便宜。因此不建议在单纯需要菌落鉴定时使用。);
◆ 方法二:直接做菌液 PCR,吸 1ul 摇好的菌液,PCR 的酶可以用做 RT-PCR 的 2×Mix,PCR 引物直接用做克隆构建用的 PCR 引物,电泳看是否有目的条带。
△ 菌液 PCR 就是利用菌体高温裂解释放出的 DNA 为模板进行的 PCR 反应,菌液 PCR 需要先挑菌落于 1ml 含抗生素 LB 中,摇菌 1-2h,然后取 0.5μl 加入到 PCR 体系中,然后上机。
△ 第一天下午质粒转化后铺板,第二天早上挑菌落,进行菌液 PCR 检测,电泳完看到结果基本上快中午了。如果此时直接把 PCR 阳性的菌落/菌液拿去摇菌,时间安排就很难受,摇 10h 是晚上,摇 16h 是半夜。所以可以将菌液 PCR 这前摇了 2 小时的菌液放到 4℃,待晚上离开实验室前,再加 LB 培养基摇菌,第二天早上来提质粒。
△ 菌液 PCR 体系:
△ PCR 程序:
95℃ 15min,时间短的话,可能解链不完全
95℃ 30s,55℃ 30s,72℃ 60s,25 个循环
72℃ 7min,暂存 4℃或电泳 -
3、测序:有条件的实验室可以自己测,没有条件的话,就送公司检测,费用不贵,每个反应十几元,每个反应能测约 700bp。这也是前面设计引物时控制 PCR 产物长度在 200-500 的重要原因之一。
质粒构建完成,送测序,推荐直接送菌液测序,公司提取后检测。自己提取质粒然后送测序,有可能会测不出来。若要自己提,推荐 OMEGA 质粒小提盒,无内毒素为佳。测序结果与目标序列对比,正确则为构建成功。 -
4、测序后抽提重组质粒(大抽),按试剂盒说明书提取,用于后续步骤。
-
推荐:OMEGA,
E.Z.N.A. Endo-Free Plasmid Mini Kit(D6948-01)
2. 转染
脂质体转染
- 参考:什么是脂质体?脂质体转染的原理和步骤?
- 1、提前铺细胞,使转染时密度达到 80%左右。密度太低细胞容易死,太高影响转染效率;
- 2、使用
OPTI-MEM分别稀释 lipofectmine 2000(脂质体载体,转染试剂) 和 重组质粒(没有内毒素的)后轻轻混匀(脂质体不耐吹打),室温孵育 5min 后逐滴加入提前铺好的待转染细胞中。 - 推荐:Lifetechnology,
Lipofectamine 2000 Reagent(11668)质粒比较大或细胞难转染,可以用 Lipofectamine 3000。
慢病毒包装(以 PLKO.1-PURO 体系为例)
● (1) 材料:psPAX2(无内毒素),pMD2.G(无内毒素),测序正确的带有插入片段的 PLKO.1-PURO(无内毒素),HEK-293T cells,OPTI-MEM® serum-free media,转染试剂 lipofectmin 2000。
● (2) 步骤(以 60mm 圆皿为例):
- a) 转染前一天,在 60mm 圆皿中接种 HEK-293T 细胞,37℃,5% CO2 培养过夜。注意接种时调整接种密度,使转染时细胞融合约 50-80%。如果养细胞时加了双抗,此时要去掉抗生素。
- b) 转染当天:(尽量下午或晚上转染)
△ 预混质粒,三个质粒的比例是可以优化的。
△ 预混 LIPO2000:LIPO2000 20μl 加 OPTI-MEM 至 500μl。
△ 两者混合,室温静置 5min,将混合液缓慢分散加入培养皿中,37℃,5% CO2 继续培养。
- c) 转染次日:转染 8-12h 后更换完全培养基,可恢复双抗,37℃,5% CO2 培养 24h。
- d) 转染第二日:收集培养基上清,即可得到慢病毒颗粒,1200rpm 离心 5min,去除混杂的 293T 细胞,存-80℃。
- e) 转染第三日:再次收集培养基上清,也可得到慢病毒颗粒,1200rpm 离心 5min,去除混杂的 293T 细胞,存-80℃。
- f) 病毒滴度测定及 MOI 测定:采用有限稀释法,建议参考吉凯、吉玛、汉恒公司慢病毒使用手册。
3. 检测过表达效果
- QPCR 或 WB 检测
- 详见:
【实验技术笔记】RNA 抽提 + 反转录 PCR + PCR 引物设计 + RT-qPCR
【实验技术笔记】Western Blotting 实验操作要点及数据分析
附表:常用酶切位点保护碱基



